Assiduviridae
| Assiduviridae | ||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
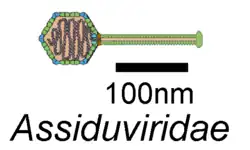
Schemazeichnung | ||||||||||||||
| Systematik | ||||||||||||||
| ||||||||||||||
| Taxonomische Merkmale | ||||||||||||||
| ||||||||||||||
| Wissenschaftlicher Name | ||||||||||||||
| Assiduviridae | ||||||||||||||
| Links | ||||||||||||||
|
Assiduviridae ist eine im Jahr 2022 als Familie eingerichtete Gruppe von Viren mit Kopf-Schwanz-Aufbau vom Morphotyp B (Siphoviren). Ihre Wirte sind Bakterien der Gattung Cellulophaga (Flavobacteriia), was sie als Bakteriophagen, speziell Flavophagen, klassifiziert.[1][2][A. 1]
Beschreibung
Die Familie Assiduviridae wurde (zusammen mit anderen) aufgrund eines Vorschlags eingerichtet, der darauf abzielte, einigen zu diesem Zeitpunkt neuen bzw. noch nicht klassifizierten Viren, die als sog. Flavophagen Mitglieder der Bakterien-Klasse Flavobacteriia infizieren, in die offizielle taxonomische Systematik aufzunehmen. Die Familie umfasst als Gründungsmitglieder die drei Gattungen Nekkelsvirus, Cebadecemvirus und Cellubavirus, die jeweils monotypisch sind mit den Arten Nekkelsvirus Nekkels, Cebadecemvirus phi10una respektive Cellubavirus phi19una. Die Isolierung und Charakterisierung dieser neuen Flavophagen sowie die Suche nach verwandten Phagen aus Sequenzdatenbanken war zuvor in Nina Bartlau et al. (2022) beschrieben.[3] Die meisten der Phagen wurden aus Oberflächenmeerwasser isoliert, das während der Phytoplanktonblüten im Frühjahr von der ökologischen Langzeitforschungsstation Helgoland Reede (englisch Helgoland Roads) beprobt wurde.[4][5][6] Die hier vorgeschlagenen taxonomischen Zuordnungen beruhen auf fünf verschiedenen Methoden:[4]
- Nukleotid-basierte Ähnlichkeiten zwischen der verschiedenen Genomen, berechnet per VIRIDIC Web-Service[7]
- Protein-basiertes hierarchische Cluster-Analyse, berechnet mit VirClust[8]
- Aminosäure-basierte phylogenetische Bäume basierend auf dem Ganzgenom, berechnet mit Viral Proteomic Tree (ViPTree)
- Aminosäure-basierte phylogenetische Bäume basierend auf dem Ganzgenom, berechnet mit der Virus Classification and Tree Building Online Resource (VICTOR)
- Kernprotein-basierte Bäume mit IQ-Trees
Zur Berechnung nukleotidbasierter Ähnlichkeiten zwischen den jeweiligen Genomen wurde VIRIDIC verwendet. Die innere Systematik der Familie(n) wurde durch Berechnung proteinbasierter hierarchischer Clustering-Bäume (der Kernproteine - englisch core proteins) mittels VirClust, MUSCLE, SH-aLRT und ModelFinder ermittelt. Zu den 32 gemeinsamen Kerngenen der Gründungsmitglieder gehören u. a. eine DNA-Primase/Helikase, eine Pektatlyase[9][10] und ein Strukturprotein.[4]
Cellulophaga-Phage Nekkels_1 als exemplarisches Virus (Phage) der Spezies Nekkelsvirus Nekkels wurde aus Oberflächenwasser vor Helgolands Dünenhafen geprobt, sein Wirt ist Cellulophaga sp. HaHa_2_1 (DSM 111038) aus der Bakterienfamilie Flavobacteriaceae. Stichprobentests zeigten, dass diese Phagenspezies während des größten Teils des Probenahmezeitraums vorhanden ist. Ein zweiter Phage, Nekkels_2, wies eine Nukleotidähnlichkeit von 97,1 % auf. Der Kapsid-Durchmesser beträgt 54,8 ± 5,0 nm, der Schwanz hat eine Länge von 141,0 ± 7,9 nm und einen Querschnittsdurchmesser von13,0 ± 1,5 nm. Die Genome beider Phagen unterschieden sich in ihrer Länge: Nekkels_2 kodiert in seinem 54.332 bp umfassenden Genom zwei Offene Leserahmen (ORFs) mehr als Nekkels_1 mit 53.385 bp. Beide zirkulären Genome haben einen GC-Gehalt von 31,5 %. Die Genome kodieren u. a. ein Hauptkapsidprotein (MCP), Halsprotein, Terminase, Pektatlyase, ein Lysozym (GH19) und ein potenzielles Spanin-Protein.[11] Die Sequenzähnlichkeit der Nekkels-Phagen mit Cellulophaga-Phage phi19:1 (Cellubavirus phi19una) beträgt 40,5 %. Wegen des langen Schwanzteils werden die Nekkels-Phagen zum Morphotyp der Siphoviren klassifiziert. Nekkels ist auch in der Lage einen anderen Wirt aus der Familie Flavobacteriaceae zu infizieren, nämlich Polaribacter sp. AHE13PA [P. sp. AHE53.11, DSM 111061], jedoch mit geringerer Effizienz.[12][13][4]
Cellulophaga-Phage phi10:1 (ϕ10:1) als exemplarischer Phage von Cebadecemvirus phi10una wurde mit Wirt Cellulophaga sp. #10 im Jahr 2000 aus der Öresund-Meerenge isoliert. Es hatte ebenfalls eine siphovirale Morphologie, seine Genomgröße beträgt 53.664 bp.[14][4]
Cellulophaga-Phage phi19:1 (ϕ19:1) schließlich als exemplarischer Phage von Cellubavirus phi19una wurde mit Wirt Cellulophaga sp. #19 im Jahr 2000 isoliert ebenfalls von dort isoliert. Auch er hatte eine siphovirale Morphologie bei einer Genomgröße von 57.447 bp.[14][4]
Systematik
Die Systematik der Assiduviridae ist nach International Committee on Taxonomy of Viruses (ICTV), ergänzt um Vorschläge nach der Taxonomie des National Center for Biotechnology Information (NCBI) in Hochkommata mit Stand 29. April 2025 (MSL#40v1) wie folgt:[1][15][16]
Familie Assiduviridae
- ohne zugewiesene Unterfamilie
- Gattung Cebadecemvirus
- Spezies Cebadecemvirus phi10una, mit
- Cellulophaga-Phage phi10:1 (ϕ10:1)[14]
- – Wirt: Cellulophaga baltica #10 [Cellulophaga sp. #10]
- – Fundort: Dänemark: Öresund, südliches Kattegat, 1 m Tiefe
- Spezies Cebadecemvirus phi10una, mit
- Gattung Cellubavirus
- Spezies Cellubavirus phi19una, mit
- Cellulophaga-Phage phi19:1 (ϕ19:1)[14]
- – Wirt: Cellulophaga baltica #19 [Cellulophaga sp. #19]
- – Fundort: Dänemark: Öresund, südliches Kattegat, 1 m Tiefe
- Spezies Cellubavirus phi19una, mit
- Gattung Nekkelsvirus
- Spezies Nekkelsvirus Nekkels, mit
- – Wirt: Cellulophaga sp. HaHa_2_1
- – Fundort: SW vom Dünenhafen, Insel Düne, Helgoland, Nordsee; 3. Mai 2018
- Spezies „Cellulophaga-Phage Nekkels_2“, mit
- Phage Nekkels_2[3]
- – Wirt: Cellulophaga sp. HaHa_2_1
- – Fundort: wie oben; 3. April 2018
- Spezies Nekkelsvirus Nekkels, mit
- Gattung Cebadecemvirus
Die Akronyme folgen Karin Holmfeldt et al. (2013).[14]
Namensherkunft (Etymologie)
Der Name der Familie Assiduviridae kommt von lateinisch assiduus ‚beständig‘ oder ‚regelmäßig‘, dies bezieht sich auf die regelmäßige Isolierung der Mitglieder der Familie aus Anreicherungskulturen von Meerwasser; der Suffix ’-viridae kennzeichnet Virusfamilien.[2][4]
Die Bezeichnung der Gattung Cebadecemvirus ist als Kofferwort nach ihrem Wirt Cellulophaga baltica benannt, der Mittelteil ist lateinisch decem ‚zehn‘ verweist auf den Stamm #10. Das Art-Epitheton phi10una verweist auf den exemplarischen Phagen phi10:1 (ϕ10:1), lateinisch una ‚eins‘ (feminin) verweist auf den ersten identifizierten Phagen des Cellulophaga -Stamms #10.[4]
Auch die Bezeichnung der Gattung Cellubavirus ist als Kofferwort nach ihrem Wirt Cellulophaga baltica benannt. Das Art-Epitheton phi19una verweist analog wieder auf den exemplarischen Phagen phi19:1 (ϕ19:1), lateinisch una ‚eins‘ (feminin) verweist auf den ersten identifizierten Phagen des Cellulophaga -Stamms #19.[4]
Die Gattung Nekkelsvirus und das Art-Epitheton Nekkels verweisen auf den exemplarischen Phagen Nekkels_1. Nekkels ist ein Vorname friesischen Ursprungs, der auf Helgoland gesprochenen Sprache, wo der Phage in den umgebenden Meeresgewässern gefunden wurde.[4]
Anmerkungen
- ↑ Die Bezeichnung Flavophagen für die Viren der Flavobacteriia ist nicht zu verwechseln mit den Flaviviren, einer umgangssprachlichen Bezeichnung für die ssRNA-Viren der Familie Flaviviridae (und speziell der Gattung Orthoflavivirus – früher Flavivirus), zu der u. a. der Erreger des Gelbfiebers gehört.
Einzelnachweise
- ↑ a b ICTV: MSL #40.v1, 3. März 2025.
- ↑ a b ICTV: Family: Assiduviridae.
- ↑ a b c Nina Bartlau, Antje Wichels, Georg Krohne, Evelien M. Adriaenssens, Anneke Heins, Bernhard M Fuchs, Rudolf Amann, Cristina Moraru: Highly diverse flavobacterial phages isolated from North Sea spring blooms. In: Oxford Academic: The ISME Journal, Band 16, Nr. 2, Februar 2022, S. 555–568; doi:10.1038/s41396-021-01097-4, PMC 8776804 (freier Volltext), PMID 34475519, Epub 2. September 2021 (englisch).
- ↑ a b c d e f g h i j Nina Bartlau, Liliana Cristina Moraru, Antje Wichels, Karin Holmfeldt, Evelien M. Adriaenssens, Rudolf Amann: Create nine new families (Pachyviridae, Pervagoviridae, Assiduviridae, Helgolandviridae, Duneviridae, Molycolviridae, Winoviridae, Forsetiviridae, and Aggregaviridae) including 13 new genera and 18 new species (Caudoviricetes). Vorschlag 2021.029B (zip:docx), Filelist.
- ↑ Hanno Teeling et al.: Helgoland Roads. Spring blooms at Helgoland. Max-Planck-Institut für Marine Mikrobiologie, Abteilung Molekulare Ökologie (mpi-bremen.de).
- ↑ Inga Kirstein et al.: AG Langzeitbeobachtungen (LTO). Alfred-Wegener-Institut (awi.de).
- ↑ VIRIDIC Web-Service (viridic.icbm.de).
- ↑ VirClust (virclust.icbm.de).
- ↑ Wikidata: Pectic acid lyase (Pectate lyase, Q24776974).
- ↑ EC 4.2.2.2. Auf Expasy (expasy.org).
- ↑ SIB: Holin/endolysin/spanin cell lysis. Auf: ViralZone (Expasy).
- ↑ Polaribacter sp. AHE53.11, AHE13PA, DSM 111061. Auf: BacDive (dsmz.de).
- ↑ NCBI Taxonomy Browser: Polaribacter sp. AHE13PA (species). Nucleotide: Polaribacter sp. AHE13PA chromosome, complete genome, Accession: CP058985.
- ↑ a b c d e Karin Holmfeldt, Natalie Solonenko, Manesh Shah, Kristen Corrier, Lasse Riemann, Nathan C. Verberkmoes, Matthew B. Sullivan: Twelve previously unknown phage genera are ubiquitous in global oceans, In: PNAS, Band 110, Nr. 31, 30. Juli 2013, S. 12798–12803; doi:10.1073/pnas.1305956110, PMC 3732932 (freier Volltext), PMID 23858439, ResearchGate:249649168, Epub 15. Juli 2013 (englisch).
- ↑ ICTV: Master Species Lists (MSL).
- ↑ NCBI Taxonomy Browser: Assiduviridae, Details: Assiduviridae (family); Nucleotide: txid2946156[Organism:exp] , Assiduviridae.
- ↑ DSMZ: Cellulophaga phage Nekkels_1 (Nekkelsvirus Nekkels) DSM 111239.